Fernanda Garcia-Fossa & Anne Carpenter
Em um experimento típico de microscopia quantitativa, os biólogos escolhem biomarcadores fluorescentes e medem características particulares (isto é, “métricas”) que eles supõem que serão perturbadas em suas amostras.
Mas no perfil baseado em imagem, você prefere deixar as células dizerem o que está acontecendo: a ideia é usar marcadores celulares generalizados (geralmente usando um ensaio como o Cell Painting (Bray et al. 2016) e medir todas as características morfológicas possíveis – a coleção de características ou atributos é chamada de “perfil fenotípico” e, normalmente, você mede cerca de mil atributos por célula. Em seguida, você pode analisar se alguma característica está alterada ou pode organizar as amostras em grupos com base em seus perfis baseados em imagens.
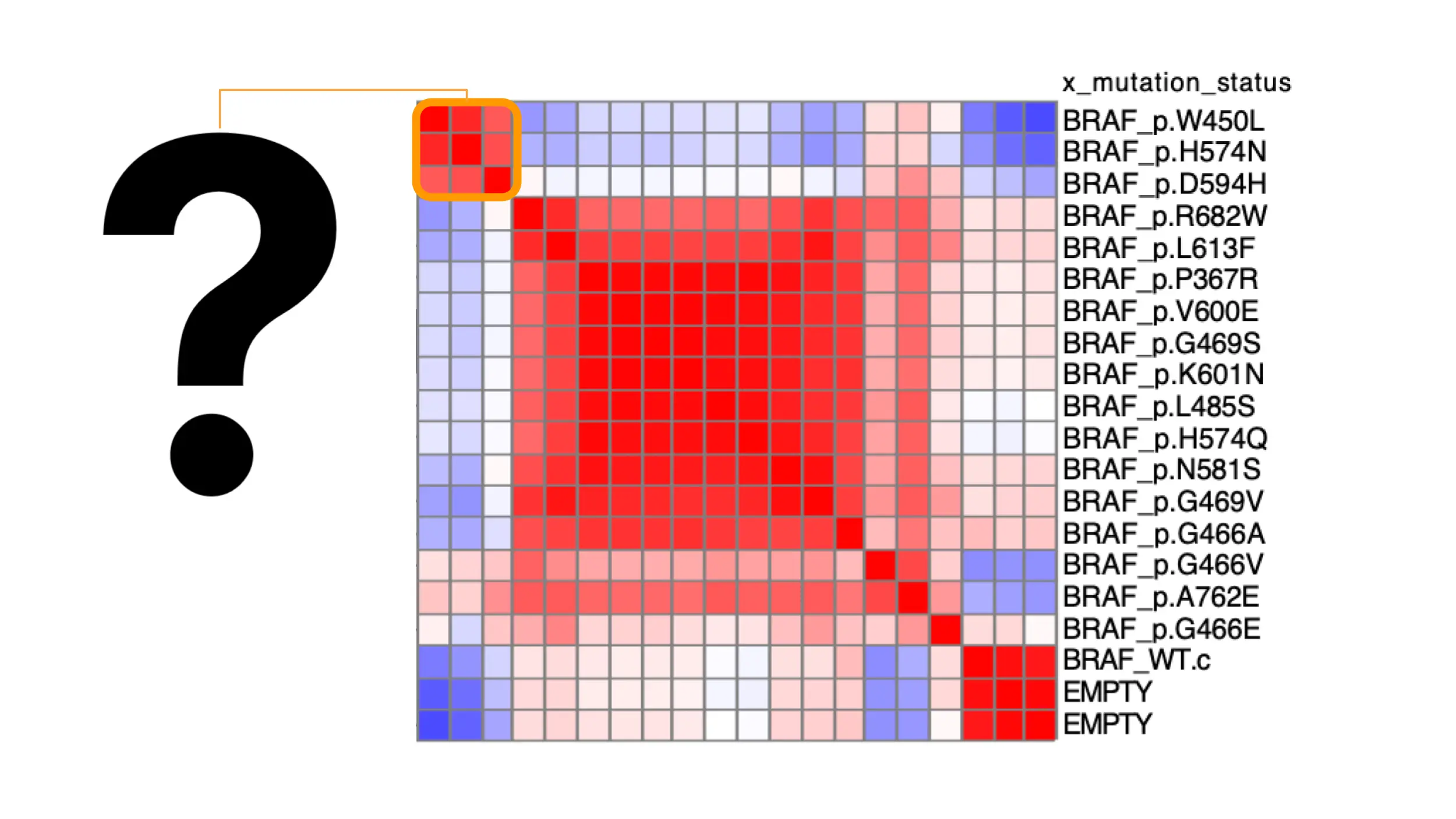
Isso leva a um gargalo comum: como você interpreta o que um determinado perfil (de uma amostra ou de um agrupamento de amostras) significa? Aqui está o um resumo aproximado que seguimos:
1. Nomes das características. Às vezes você dá sorte e o perfil é evidente em apenas uma ou algumas características avaliadas e os nomes das características têm significados super óbvios (área do núcleo ou quantidade de determinada marcação em um determinado compartimento da célula). Pode ser necessário pesquisar o significado dos nomes das características no manual do CellProfiler para entendê-los melhor e discernir sua conexão com o significado biológico. Alguns cuidados são necessários aqui; por exemplo, moléculas que causam danos ao DNA geralmente mostram características na marcação de actina como as mais dramaticamente impactadas (porque talvez as células se soltem da placa e comecem a se arredondar), então pode não haver uma relação direta entre o nome de uma característica e uma função biológica. Quer saber como você procura quais recursos são incomuns em sua(s) amostra(s)? Costumamos usar o software de código aberto Morpheus seguindo esse breve tutorial aqui (a página principal do repositório GitHub também possui links para uma versão em vídeo). Também descrevemos um protocolo de como ele é usado em nosso artigo recente.
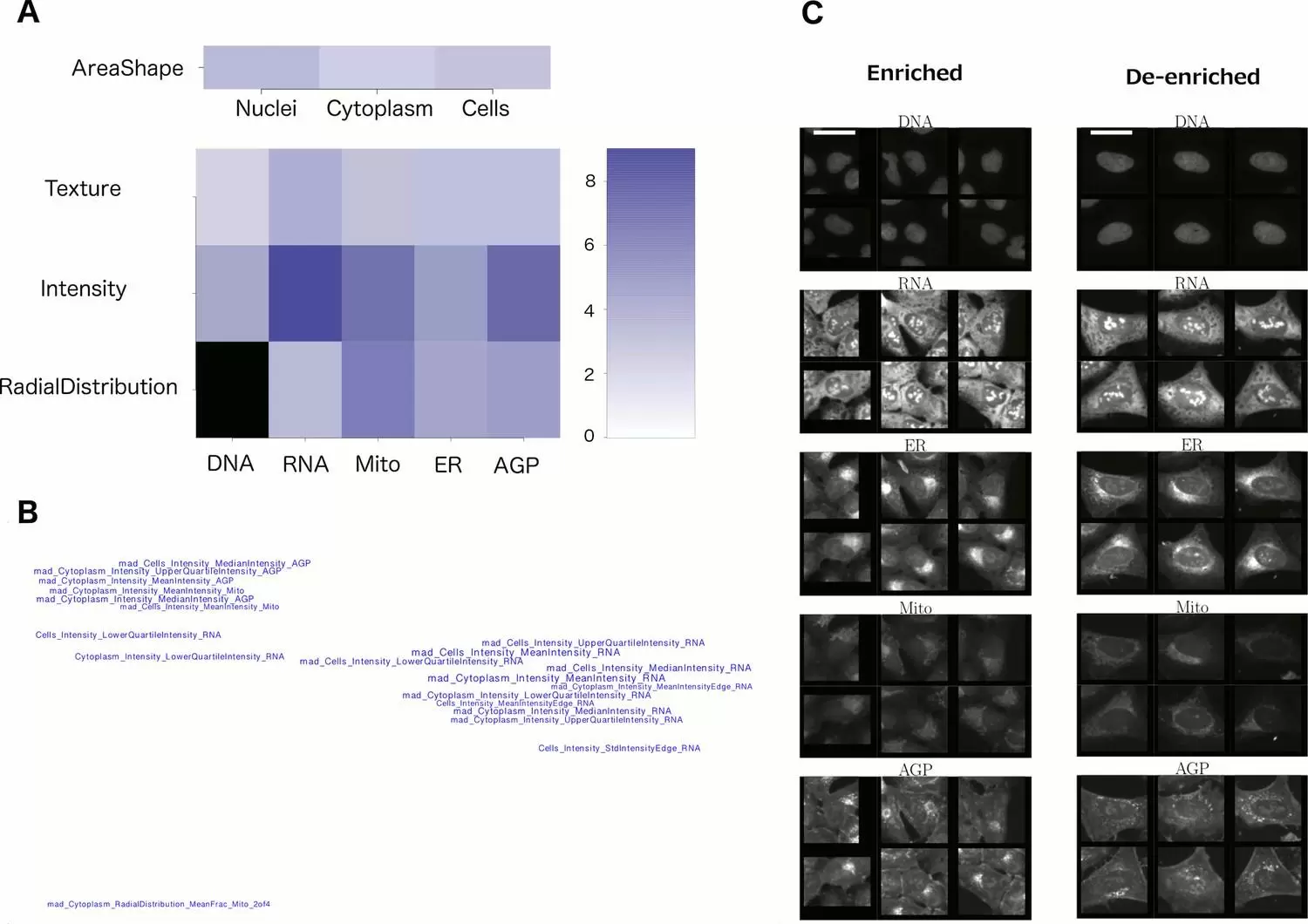
2. Categorias dos atributos. Às vezes, observar os grupos de características incomuns no perfil pode revelar um padrão útil. Conforme mostrado na Figura 2A, pode-se entender quais canais e categorias de atributos são mais influentes. Em uma visualização como a Figura 2B, onde os nomes dos atributos são organizados com base em suas semelhanças uns com os outros (correlações), percebe-se rapidamente que existem três atributos básicos influenciando esse fenótipo (em vez de ser sobrecarregado pela lista original de 20 características). Esses gráficos são criados usando a linha de comando, com scripts deste artigo.
3. Interpretação especializada. Às vezes, há muitos atributos dominantes no perfil e seu significado coletivo não é óbvio, mas um especialista pode examinar a lista e levantar algumas hipóteses. Por exemplo, uma das situações mais comuns é quando o tamanho da célula é claramente alterado – um especialista que pensa sobre características morfológicas pode rapidamente discernir a presença de várias características aparentemente não relacionadas que provavelmente são fortemente influenciadas pelo tamanho da célula, como correlações entre a marcação de outras organelas (porque as células que se tornaram pequenas e arredondadas tendem a ter mais marcações sobrepostas). Se você fez uma tentativa sólida e precisa da ajuda de um especialista, considere postar sua dúvida em forum.image.sc junto com alguns exemplos e imagens de controle e talvez alguém possa ajudá-lo.
4. Observe as imagens como um todo. Às vezes, examinar imagens de exemplo pode decifrar um perfil complexo. Você pode ficar perplexo com o que significa o eixo menor do DNA ser baixo, a área da célula ser baixa e a intensidade do DNA ser alta, mas uma rápida olhada revela que as células em questão estão no estágio de metáfase da mitose. e de repente as mudanças de recursos fazem sentido. Certifique-se de examinar imagens de amostras de controle para comparação.
5. Observe imagens de células aleatórias. Às vezes, olhar para campos como um todo é inconclusivo porque há muita heterogeneidade entre as células na imagem. Neste caso, pode ser útil examinar células individuais. Descrevemos scripts para exibir células aleatórias de uma população (juntamente com controles negativos) em nosso recente protocolo.
6. Veja imagens representativas de uma única célula. Células únicas aleatórias ainda podem refletir heterogeneidade substancial, o que é confuso; pode ser útil exibir subpopulações específicas de células. Existem duas maneiras que recomendamos: (a) Exibir células cujas medidas estão mais próximas da média da população. Novamente, nosso protocolo recente tem scripts para realizar isso e um protocolo passo a passo para fazê-lo. (b) Determinar computacionalmente quais fenótipos de células são enriquecidos e enriquecidos para sua(s) amostra(s) de interesse. Esta não é uma etapa trivial, mas às vezes é crítica para entender a mudança morfológica. Por exemplo, na imagem mostrada na Figura 2C, fica claro (junto com o conhecimento de quais características procurar, das Figuras 2B e 2C), que as células enriquecidas à direita tendem a ser grandes e planas, com organelas relativamente simétricas. Essas exibições são criadas usando a linha de comando, com scripts deste artigo.
7. Correspondência a outros perfis anotados. Às vezes, o perfil pode “combinar” ou “opor-se” a perfis de outras amostras que possuem anotações (um perfil fenotípico que é conhecido, i.e. um composto que é descrito na literatura alterando o tamanho do núcleo) e essas anotações fornecem insights. Isso requer (a) uma fonte de dados de compostos anotados e/ou genes em um modelo de célula semelhante e um conjunto de marcadores fluorescentes, para consulta/comparação, (b) uma alta similaridade entre os perfis de seu experimento e do banco de dados disponível, e (c) habilidades computacionais para realizar as consultas, visto que atualmente não há ferramentas fáceis de usar para realizar a identificação de amostras de alta correlação e anticorrelação. Nós e outros grupos estamos trabalhando para criar ferramentas que esperamos que tornem isso mais fácil para a comunidade nos próximos anos; os scripts para essas etapas estão disponíveis em nesse artigo recente. O software comercial também está começando a oferecer essa opção. Ardigen criou um portal de dados gratuito que acessa os dados públicos da JUMP Cell Painting, onde é possível baixar listas de amostras “próximas” (fenotipicamente semelhantes) a uma amostra de interesse. O Spring Discovery também criou um portal de dados gratuito que acessa os dados públicos do JUMP Cell Painting que em breve oferecerão seus recursos de MegaMap para combinar amostras. O uso de software em dados internos ou privados requer contato com as empresas para obter mais informações.
8. Associar à perturbação. Às vezes, você não consegue progredir muito na compreensão real do fenótipo induzido, então você apenas deixa suas perturbações específicas do projeto dizerem o que está acontecendo (ou seja, adicionamos essa droga, que sabemos que faz X, e vemos essa nova população de células aparecer, então chamamos essas células de “células induzidas por X”).
Interpretar perfis de morfologia complexos pode ser um desafio, mas vale a pena o esforço quando você descobre algo novo!
Referencias:
- Bray MA, Singh S, Han H, Davis CT, Borgeson B, Hartland C, Kost-Alimova M, Gustafsdottir SM, Gibson CC, Carpenter AE. Cell Painting, a high-content image-based assay for morphological profiling using multiplexed fluorescent dyes. Nat Protoc. 2016 Sep;11(9):1757–1774. PMCID: PMC5223290